За последнее десятилетие ученые адаптировали системы CRISPR от микробов к технологии редактирования генов, точной и программируемой системе для модификации ДНК. Теперь ученые из Института исследований мозга Макговерна при Массачусетском технологическом институте и Института Броуд при Массачусетском технологическом институте и Гарварде открыли новый класс программируемых систем модификации ДНК, называемых OMEGA (обязательная активность, управляемая мобильными элементами), которые, естественно, могут участвовать в перетасовке небольших фрагментов. ДНК во всех бактериальных геномах.
Эти древние ферменты, расщепляющие ДНК, направляются к своим целям с помощью небольших кусочков РНК. Хотя они возникли в бактериях, теперь они созданы для работы в клетках человека, что позволяет предположить, что они могут быть полезны при разработке методов лечения, редактирующих гены, особенно потому, что они маленькие (около 30 процентов от размера Cas9), что упрощает их использование, доставляют в клетки, чем более объемные ферменты. Открытие, опубликованное в журнале Science, свидетельствует о том, что природные ферменты, управляемые РНК, являются одними из самых распространенных белков на Земле, что указывает на обширную новую область биологии, которая готова совершить следующую революцию в геноме. технология редактирования.
Программируемые ферменты, особенно те, которые используют руководство по РНК, можно быстро адаптировать для различных целей. Например, ферменты CRISPR естественным образом используют руководство по РНК для нацеливания на вирусных захватчиков, но биологи могут направить Cas9 на любую цель, создав свои собственные руководства по РНК.
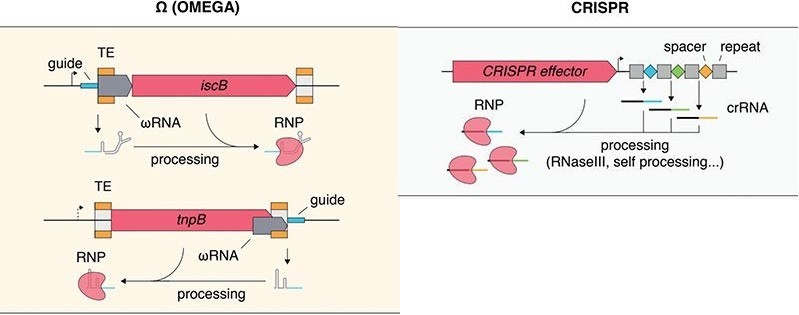
Первые намеки на то, что белки OMEGA могут управляться РНК, пришли из генов белков, называемых IscB. IscB не участвуют в иммунитете CRISPR и, как известно, не связываются с РНК, но выглядят как небольшие ферменты, разрезающие ДНК. Команда обнаружила, что рядом с каждым IscB закодирована небольшая РНК, и она заставляла ферменты IscB разрезать определенные последовательности ДНК. Они назвали эти РНК "ωРНК".
Эксперименты команды показали, что два других класса небольших белков, известные как IsrBs и TnpBs, одни из самых распространенных генов у бактерий, также используют ωРНК, которые действуют как направляющие для управления расщеплением ДНК.
IscB, IsrB и TnpB находятся в мобильных генетических элементах, называемых транспозонами. Хан Алтае-Тран, аспирант Массачусетского технологического института в области биологической инженерии и соавтор статьи, объясняет, что каждый раз, когда эти транспозоны перемещаются, они создают новую направляющую РНК, позволяя ферменту, который они кодируют, разрезать где-то еще.
Неясно, как бактерии получают выгоду от этого перетасовки генома, и приносят ли они вообще пользу. По словам авторов, транспозоны часто рассматриваются как эгоистичные кусочки ДНК, озабоченные только своей собственной подвижностью и сохранностью. Но если хосты могут "кооптировать" эти системы и перепрофилировать их, хосты могут получить новые возможности, как в случае с системами CRISPR, которые обеспечивают адаптивный иммунитет.
IscB и TnpB являются предшественниками систем Cas9 и Cas12 CRISPR. Команда подозревает, что они вместе с IsrB, вероятно, дали начало и другим РНК-управляемым ферментам, и они очень хотят их найти. По словам ученых, их интересует спектр функций, которые могут выполняться в природе ферментами, управляемыми РНК, и подозревают, что эволюция, вероятно, уже использовала ферменты OMEGA, такие как IscB и TnpB, для решения проблем, с которыми биологи стремятся решить.
Команда также заинтересована в отслеживании эволюции систем, управляемых РНК, в далеком прошлом. Ученые считают, что понимание истоков этих процессов может проложить путь к разработке еще большего числа классов программируемых инструментов.